Práctica 15 Aislamiento de DNA PDF

| Title | Práctica 15 Aislamiento de DNA |
|---|---|
| Author | Oscar Espinosa Alvarez |
| Course | Bioquímica |
| Institution | Instituto Politécnico Nacional |
| Pages | 7 |
| File Size | 390.1 KB |
| File Type | |
| Total Downloads | 160 |
| Total Views | 667 |
Summary
IntroducciónEl ácido desoxirribonucleico (DNA) es una molécula muy larga que contiene muchos miles de desoxirribonucleicos de cuatro clases distintas, unidos en una secuencia que es característica para cada organismo y que consta, generalmente de dos cadenas.Las funciones del ADN consisten en almace...
Description
Instituto Politécnico Nacional Escuela Nacional de Ciencias Biológicas Bioquímica General
Práctica 15 “Aislamiento de DNA de espinaca o guayaba, hidrolisis de RNA y DNA e identificación de bases púricas y pirimídicas por cromatografía en capa fina”
Integrantes: -Espinosa Alvarez Oscar -Pérez Ramírez Alexandra Itzel.
Introducción El ácido desoxirribonucleico (DNA) es una molécula muy larga que contiene muchos miles de desoxirribonucleicos de cuatro clases distintas, unidos en una secuencia que es característica para cada organismo y que consta, generalmente de dos cadenas. Las funciones del ADN consisten en almacenar la información genética completa, necesaria para especificar la estructura de todas las proteínas y en cada una de las clases de RNA del organismo, en programar, tanto en el tiempo como en el espacio, la biosíntesis ordenada de los componentes de las células y de los tejidos, en determinar la actividad de un organismo a lo largo de su ciclo vital y finalmente, en definir la individualidad de un organismo dado. Los ácidos nucleicos, al igual que las proteínas son biopolímeros de alto peso molecular constituidos por nucleótidos, los cuales se encuentran unidos entre sí por enlaces covalentes denominados fosfodiéster. Un nucleótido está formado por una base heterocíclica nitrogenada derivada de la purina o de la pirimidina, de un azúcar y de un grupo fosfato. Dependiendo del tipo de nucleótido existen dos clases de ácidos nucleicos: ácido desoxirribonucleico (DNA) y ácido ribonucleico (RNA) Las bases nitrogenadas de los ácidos nucleicos absorben fuertemente luz ultravioleta en la región 260-280 nm. La longitud de onda de máxima absorción de mezclas de las bases comunes es aproximadamente de 260 nm.
Grupo: 3FV1
Objetivo Desintegrar las bases nitrogenadas del DNA y RNA por cromatografía, así como reconocerlos por la propiedad que tienen de adsorber luz UV.
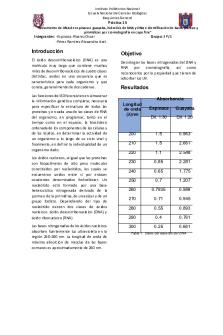
Resultados Absorbancia Longitud de onda (λ)nm
Espinaca
Guayaba
Dil. 1:30
Dil. 1:20
200
1.5
0.963
210
1.5
2.661
220
1.1
2.548
230
0.85
2.281
240
0.65
1.775
250
0.7
1.207
260
0.7935
0.989
270
0.71
0.945
280
0.55
0.893
290
0.4
0.761
300
0.25
0.661
Tabla 1. Datos del espectro del DNA
Absorbancia
Espectro UV del DNA de la espinaca 1.6 1.4 1.2 1 0.8 0.6 0.4 0.2 0 190
210
230
250
270
290
310
290
310
Longitud de onda (λ)nm
Grafica 1. Espectro UV del DNA de la espinaca
Espectro UV del DNA de la guayaba 3
Absorbancia
2.5 2 1.5 1 0.5 0 190
210
230
250
270
Longitud de onda (λ)nm
Grafica 2. Espectro UV del DNA de la guayaba DNA de espinaca
DNA de guayaba
Pureza= 0.55 =1.4427 100% pureza= 2.0 X2--------1.4427 X2 pureza = 72.13%
0.7935
Pureza= 0.893 =1.1075 100% pureza= 2.0 X2--------1.1075 X2 pureza = 55.37%
Nuestro DNA de espinaca está contaminado con proteínas
Nuestro DNA de guayaba está contaminado con proteínas
0.989
Tabla 2. Pureza del DNA de espinaca y guayaba
Nomograma
Espinaca
Guayaba
0.825mg/mL
0.6mg/mL
Fórmula utilizando la absortividad molar del DNA puro 1.058mg/mL 0.879mg/mL Relación utilizada en estudios de biología molecular
1.19mg/mL
0.989mg/mL
Pureza
72%
55%
Proteínas
7.5mg/mL
13 mg/mL
Tabla 3. Concentraciones del DNA por los 3 métodos, pureza y concentración de proteínas
Método 1. Nomograma
. C=0.0275mg/mL (30) =0.825 Espinaca C=0.030mg/mL (20) =0.6 Guayaba C=0.25(30) =7.5 mg/mL C=0.65(20) =13 mg/mL
2. Fórmula utilizando la absortividad molar del DNA puro C=(As260/22500) x factor de dilución Fórmula g/mL Espinaca C= (0.7935/22500) x30=1.058x10-3g/mL=1.058mg/mL Guayaba C= (0.989/22500) X20=3.79x10-4g/mL=0.879mg/mL
3.Relación utilizada en estudios de biología molecular (260nm)1.0 Abs--------50µg/mL Fórmula µg/mL 260 calculada-----------” a” C=a x factor de dilución Espinaca C= (0.7935) (50 µg/mL) (30) =1190.25µg/mL=1.19mg/mL Guayaba C= (0.989) (50 µg/mL) (20) = 989µg/mL=0.989mg/mL
Hidrólisis de RNA y DNA e identificación de bases púricas y pirimídicas por cromatografía en capa fina
Rf=
𝑑𝑅
𝑑𝐹𝑀
Rf=
𝑑𝑖𝑠𝑡𝑎𝑛𝑐𝑖𝑎 𝑑𝑒𝑙 𝑖𝑛𝑖𝑐𝑖𝑜 𝑎𝑙 𝑐𝑒𝑛𝑡𝑟𝑜 𝑑𝑒 𝑙𝑎 𝑚𝑎
𝑑𝑖𝑠𝑡𝑎𝑛𝑐𝑖𝑎 𝑑𝑒𝑙 𝑖𝑛𝑖𝑐𝑖𝑜 ℎ𝑎𝑠𝑡𝑎 𝑒𝑙 𝑓𝑟𝑒𝑛𝑡𝑒 𝑑𝑒𝑙 𝑑𝑖𝑠𝑜𝑙𝑣𝑒𝑛𝑡𝑒
dFM= 11 cm Bases nitrogenadas presentes en Muestra Tiamina Adenina Guanina Citosina Uracilo
Rf 7.67 11 5.05
=0.6972
DNA ✔
RNA --
=0.4590
✔
✔
=0.2936
✔
✔
=0.1972
✔
✔
=0.6109
--
✔
11 3.23 11 2.17 11 6.72 11
Tabla 4. Rf de las bases nitrogenadas en la cromatografía en capa fina
Discusión Aislamiento del DNA de origen vegetal y su caracterización espectrofotométrica Según los resultados obtenidos Como se indica en la tabla 2, la pureza de la espinaca es de 72% y de la guayaba es de 55%, la cantidad de proteínas es mayor en la de menor pureza. En la gráfica 1 en absorbancias a 230 de la guayaba son muy altas, la pendiente de la guayaba en longitudes de 260 a 280 es menor que el de la espinaca, también indica una alta cantidad de proteínas con grupos aromáticos (histonas) Como se indica en la tabla 3, los métodos 2 y 3 son más parecidos que el nomograma, el nomograma es el más impreciso, pero nos brinda la concentración de proteínas Las histonas forman parte de la estructura terciaria del ADN, estas se eliminan parcialmente en la técnica, perdiendo la estructura y conservando únicamente la secundaria. Hidrólisis de RNA y DNA e identificación de bases púricas y pirimídicas por cromatografía en capa fina La hidrólisis de DNA y ARN se llevó a cabo haciéndolos reaccionar con HCl 6 N y sometiéndolos a condiciones de alta presión pues se introdujo la solución a hidrolizar al autoclave a 15 lb/ pulg^2 durante 12 horas en 3 ciclos de 4 horas cada uno estas condiciones son necesarias ya que, el ADN está formado de dos cadenas enrolladas alrededor de un eje formando una hélice doble, el ADN contiene bases nitrogenadas que son hidrofóbicas y están apiladas en el interior de la doble hélice, la parte exterior es hidrofóbica, constituida por grupos fosfato y azucares. Y sobre todo las bases de una de las cadenas o hebras están unidas mediante puentes de hidrogeno con las bases nitrogenadas de la otra cadena, uniendo ambas cadenas; la adenina se une a la Timina con dos puentes de hidrogeno y la guanina se une a la citosina con tres puentes de hidrogeno este es un
enlace más estable. Por lo que se requiere una presión elevada al momento de pasar por los ciclos antes mencionados también se adquirirá calor, esto favorece a que los puentes de hidrogeno se rompan para obtener la molécula hidrolizada. Ya con el hidrolizado se lleva a cabo la aplicación del cromatograma en capa fina la utilidad de esta técnica es que la fase móvil ayudara a separar los componentes del ARN y DNA a través de la migración diferencial sobre la capa fina, además de que en esta se pudo analizar varias muestras en una sola placa cromatográfica y se utilizó una pequeña cantidad de las muestras a analizar y bueno lo más importante su aplicación principal es proporcionar información sobre la homogeneidad de los componentes del ARN y DNA al ser comparados con las bases nitrogenadas puras. La fase móvil es la mezcla de butanol: ácido acético: agua (5:2:3 v/v/v) esto implica que las bases nitrogenadas cuya cadena R tenga un carácter más apolar, tendrán tendencia a desplazarse con la fase móvil, mientras que las bases nitrogenadas polares serán retenidas por la fase estacionaria (el papel utilizado está fabricado para el intercambio iónico) Y su revelación es llevada a cabo por exposición de luz ultravioleta ya que a simple vista no se percibe los Rf de cada muestra. Sin embargo, los materiales orgánicos son extremadamente sensible a este tipo de radiación; Cuando la radiación es absorbida por el material, los electrones son puestos de forma temporal en un mayor estado de energía, luego esa energía es liberada volviendo los electrones a su estado normal En la tabla de resultados podemos observar que Rf coinciden con las estructuras del ADN y ARN
CONCLUSIONES Aislamiento del DNA de origen vegetal y su caracterización espectrofotométrica •
• • • • •
La purificación del ADN involucra el rompimiento de la pared y la membrana celular, inhibición de nucleasas, precipitación del ADN impuro, disociación de proteínas del DNA y reprecipitación del DNA ya purificado. El rompimiento eficiente de la pared y membrana celular facilita la extracción del ADN Las nucleasas degradan el ADN por lo que es requerido inhibirlas. La pureza obtenida de la guayaba es menor a la espinaca. A mayor cantidad de proteínas presentes, mayor estará contaminado el producto obtenido. Los métodos 2 y 3 son más análogos y más precisos que el nomograma. El nivel estructural del DNA del producto obtenido es estructura secundaria.
Hidrólisis de RNA y DNA e identificación de bases púricas y pirimídicas por cromatografía en capa fina • • •
Los ácidos nucleicos (ARN y ARN) están constituidos por moléculas sillares en pares de purinas y pirimidinas. Las bases nitrogenadas que forman al ADN son: Adenina, timina, guanina y citosina La Adenina, timina, guanina y uracilo, constituyen al ARN.
Bibliografía • • • • •
Horton, H. (2008). “Principios de bioquímica” 4ta edición, editorial Pearson educación, México, pág. (226, 234-239). Baynes J. (2011). Bioquímica Médica. Tercera edición. España: Mosby. pág. (97-99). Alemany. M., Font, D. “Prácticas de bioquímica”, Ed. Alhambra. Madrid. pp. 285-289 (1983) Rawn, J. D. “Bioquímica”. Vol. II, Mc Graw Hill, Interamericana. Madrid. pp 665-700 (1989) Bohinski, R. C. “Bioquímica”, 5a Ed. Addison-Wesley Iberoamericana. Argentina. Brasil. pp 227-242 (1991)...
Similar Free PDFs
Práctica 15 Aislamiento de DNA
- 7 Pages

1. Aislamiento DE Tuberias
- 18 Pages

PRUEBA DE RESISTENCIA DE AISLAMIENTO
- 56 Pages
aislamiento de estreprococus
- 13 Pages

Prctica 9 historia Paula
- 3 Pages

Sistemas de aislamiento doble
- 12 Pages

Aislamiento absoluto
- 3 Pages

Nivel Aislamiento
- 1 Pages

Tema 1. AISLAMIENTO
- 7 Pages
Popular Institutions
- Tinajero National High School - Annex
- Politeknik Caltex Riau
- Yokohama City University
- SGT University
- University of Al-Qadisiyah
- Divine Word College of Vigan
- Techniek College Rotterdam
- Universidade de Santiago
- Universiti Teknologi MARA Cawangan Johor Kampus Pasir Gudang
- Poltekkes Kemenkes Yogyakarta
- Baguio City National High School
- Colegio san marcos
- preparatoria uno
- Centro de Bachillerato Tecnológico Industrial y de Servicios No. 107
- Dalian Maritime University
- Quang Trung Secondary School
- Colegio Tecnológico en Informática
- Corporación Regional de Educación Superior
- Grupo CEDVA
- Dar Al Uloom University
- Centro de Estudios Preuniversitarios de la Universidad Nacional de Ingeniería
- 上智大学
- Aakash International School, Nuna Majara
- San Felipe Neri Catholic School
- Kang Chiao International School - New Taipei City
- Misamis Occidental National High School
- Institución Educativa Escuela Normal Juan Ladrilleros
- Kolehiyo ng Pantukan
- Batanes State College
- Instituto Continental
- Sekolah Menengah Kejuruan Kesehatan Kaltara (Tarakan)
- Colegio de La Inmaculada Concepcion - Cebu






